显微镜是人类历史上的伟大发明之一,在整个显微镜的发展史上有两次重大突破,分别是光学显微镜和电子显微镜的发明。
现在,显微技术领域可能正迎来第三次革命——“DNA 显微镜”问世!
2019 年 6 月 20 日,CRISPR 基因编辑重要贡献者张锋教授及其霍华德·休斯医学研究所(HHMI)的同事,在 Cell 杂志发表重磅成果,他们开发出一种全新的 “DNA 显微镜”,可以建立细胞的图像,同时收集大量的基因组信息。
人类显微视角进入新疆域
基于光学的显微镜,可以追溯到17 世纪,它打开了人类对微观世界的认识。光学显微镜主要依靠可见光照射样本,通过一组透镜组合来放大物品。
在生物医学领域,光学显微镜的发明是一项革命性的技术突破,令我们认识到生命体的基本单位为细胞,同时大大助力人类对疾病的认知与防治,比如青霉素的发现。
之后科学家们又对光学显微方法进行了反复升级,甚至超越了可见光谱。
1913 年恩斯特·鲁斯发明的电子显微镜,更是将显示水平拉到原子级别。它让研究人员可以在原子水平去了解生理学过程及单个分子的结构。
如今,科学家已经可以使用光、x 射线和电子来观察组织和细胞内部。
大家熟悉的电子显微镜、荧光显微镜、薄层显微镜,它们都是基于探测样品发射光子或电子的原理进行观测的。通过这类显微镜,科学家们可以追踪大脑中类似丝状的神经纤维,甚至可以观察活的老鼠胚胎如何产生原始心脏的跳动细胞。
然而,这些显微镜都无法看到在基因组水平的细胞中发生了什么。
为了对核酸水平进行直接观测,实验室及临床中大多依赖分子探针技术,即将与待观察核酸互补的碱基对导入细胞中,利用碱基互补配对原则标记待测核酸,再通过荧光等其他显色物质来显示待测核酸。这种间接方法虽能令研究者观测核酸,但其标本制备过程繁琐,耗时耗力。
而此次张锋教授研发的DNA 显微技术,通过独特的成像模式,采用特殊的成像原理,可将物理图像编码 DNA,先利用标记核酸的”堆叠“编码每一种核酸,再采用数据分析”投射“其物理图像,从而实现对基因组的直接观测。

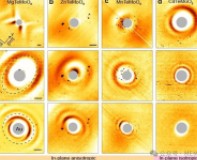
图 | DNA 显微技术显像原理
最新的DNA 显微技术显像原理与我们想象中的可能不同,并不是直接对 DNA 链进行显示。这是由于 DNA 中的每个碱基分子在每个细胞内存在的数量十分微小,哪怕我们直接标记,也很难直接观测到其标记信号。
因此,张锋教授的研究团队采用了一个十分巧妙的方法来解决这一问题,他们采用了”堆叠“分子的化学合成法。
首先,研究人员将待测细胞滴在载玻片上,并进行相应的固定。随后,往细胞内注入各种各样的DNA 标记物(这里用的是 cDNA 片段),这些 DNA 标记物会连接到与其互补的 RNA 分子上,使其具有唯一的标签。
但到此为止,我们还是不能直观地观测基因组,所以化学合成法派上用场了,研究人员以这些导入的DNA 标记物为模板,大量扩增其副本,使每一个标记核酸都挂着”一大堆标记副本“,这样通过”分子堆叠“就使的相邻的标记分子相互碰撞,进而使它们连在一起。
该研究的主要负责人之一Joshua A. Weinstein 教授表示,可以把每一个分子想象成一个向外发射自己信号的无线电发射塔。靠的越近,那么就可以产生更多的 DNA 对,”分子堆叠“效应更明显,反之,靠的越远,这些 DNA 对越少,”分子堆叠“效应更弱。
在这一过程进行大约30 个小时后,研究人员就拼凑出识别每一碱基的”分子堆“,然后该团队通过计算机算法解析这些”分子堆“信号,将原始样本中的约 5000 万个基因的碱基序列转化为图像,进而使实验者在光学显微镜下观测样本基因组信息。
参与本项研究的霍华德·休斯医学研究所研究员 Aviv Regev 表示,捕捉这样一个细胞的完整图像不需要昂贵的显微镜或很多昂贵的设备,只需要样本和一根”吸管“就够了。

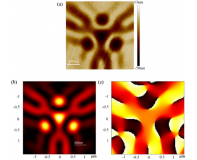
图| 序列编码显示图(其中红光为 RFP、绿光为 GFP、白光为 GAPDH)(来源:Cell)
可能引发生物医学领域的重大突破